 |
Рестрикционное картирование генов
|
|
|
|
Полную, но, к сожалению, пока трудно интерпретируемую информацию о строении гена может дать только определение его первичной структуры, т.е. последовательности составляющих ген нуклеотидов. На практике при исследовании протяженных (до 40 т.п.о.) клонированных последовательностей нуклеотидов, включающих исследуемые гены, прежде всего строят их рестрикционные карты. Рестрикционные карты представляют собой схемы, изображающие взаимное расположение сайтов рестрикции для разных рестриктаз и расстояния между ними. Поскольку каждый сайт рестрикции является не чем иным, как строго определенной последовательностью нуклеотидов ДНК, рестрикционные карты наглядно заключают в себе информацию об особенностях первичной структуры картируемых участков генома.
Для построения рестрикционной карты используют гибридизацию по методу Е. Саузерна. Клонированный фрагмент ДНК отдельно или в составе вектора получают в препаративном количестве, затем его обрабатывают соответствующими рестриктазами и продукты рестрикции разделяют электрофорезом в агарозном геле. Количество образовавшихся рестрикционных фрагментов ДНК, обнаруживаемых после окрашивания бромистым этидием в виде флуоресцирующих полос в ультрафиолетовом свете, соответствует количеству сайтов рестрикции в том случае, если различия в размерах образовавшихся фрагментов ДНК достаточны для их разделения при электрофорезе.
Размеры рестрикционных фрагментов оценивают путем сравнения их электрофоретической подвижности с таковой фрагментов ДНК известных размеров. Получив информацию о количестве сайтов рестрикции в гене, далее определяют их взаимное расположение. Для этого в качестве зондов выбирают короткие фрагменты ДНК и после введения в них радиоактивной метки их гибридизуют с рестрикционными фрагментами ДНК, которые после электрофоретического разделения в агарозном геле были перенесены на нитроцеллюлозные или нейлоновые фильтры. По завершении гибридизации положение фрагментов ДНК, связавших метку, на фильтрах обнаруживают с помощью авторадиографии. Получение такой информации о принадлежности конкретных фрагментов ДНК, образовавшихся под действием различных рестриктаз, к 5’- или 3’-концевым частям исследуемой последовательности нуклеотидов обычно бывает достаточным для определения взаимного расположения различных сайтов рестрикции на рестрикционных картах.
|
|
|
7.6.2. "Прогулки и прыжки по хромосомам"
Многие гены в геноме эукариот располагаются в виде кластеров, члены которых функционально или эволюционно связаны друг с другом. В связи с этим в молекулярной генетике часто возникает задача клонирования генов, расположенных по соседству с уже выделенными генами. Для ее решения был разработан метод " прогулки по хромосоме ". Из клонотеки генов, полученной на основе фаговых или космидных векторов, выделяют ген и короткие 5’- и 3’-концевой фрагменты его последовательности используют в качестве зондов для поиска фрагментов ДНК, перекрывающихся с этим геном. Основным требованием, предъявляемым к зонду, является принадлежность его к уникальной последовательности нуклеотидов, т.е. встречающейся в геноме только один раз. С использованием такого простого подхода были исследованы многие кластеры эукариотических генов, в частности b-глобинового локуса человека, структура которого представлена на рис. II.14.
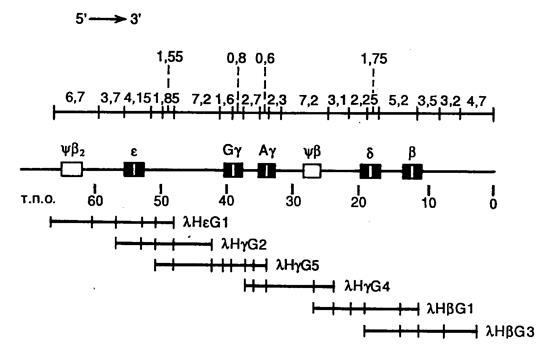
Рис. II.14. Генетическая карта локуса b-глобинового гена человека, полученная методом "прогулки по хромосоме"
Перекрывающиеся фрагменты геномной ДНК человека получены путем ее частичного расщепления рестриктазой EcoR I, сайты рестрикции для которой отмечены поперечными черточками. Указаны расстояния между EcoR I-сайтами рестрикции в т.п.о., гены b-глобинового локуса, а также названия рекомбинантных фагов l
|
|
|
С помощью "прогулки по хромосоме" удается детально картировать участки ДНК длиной до 250 т.п.о. Недавно разработанный метод "прыжков по хромосоме" позволяет стыковать за один прием фрагменты ДНК общей длиной от 100 до 500–1000 т.п.о. В этом случае на первом этапе препарат высокомолекулярной ДНК расщепляют соответствующей рестриктазой и с помощью электрофореза в импульсном электрическом поле получают фракцию фрагментов ДНК одинакового размера (в данном примере ~100 т.п.о.) (см. рис. II.15). Затем фрагменты ДНК лигируют с маркерным геном, например геном устойчивости к антибиотикам, что приводит к образованию кольцевых молекул. Кольцевые молекулы ДНК расщепляют другой рестриктазой, для которой отсутствуют сайты рестрикции в маркерном гене, что сопровождается образованием коротких фрагментов ДНК, которые далее можно клонировать обычными способами. Полученную клонотеку фрагментов ДНК исследуют с использованием гибридизации с зондом, комплементарным точке начала "прыжка по хромосоме". Отобранные в результате гибридизации клоны содержат маркерный ген, фланкированный изучаемыми последовательностями нуклеотидов, удаленными друг от друга на 100 т.п.о. Таким образом, получают информацию о последовательностях нуклеотидов, удаленных от точки начала "прыжка по хромосоме" на 100 т.п.о., и эти последовательности далее используют в качестве зонда для выделения и исследования фланкирующих ее фрагментов ДНК.

Рис. II.15. Схема метода "прыжков по хромосоме"
S1-картирование РНК и ДНК
Нуклеаза S1, специфически гидролизующая одноцепочечные ДНК и РНК, успешно используется для исследования колинеарности ДНК и кодируемой ей РНК, точного картирования мест инициации и терминации транскрипции на ДНК-матрицах, а также для оценки гомологии между двумя молекулами ДНК или РНК. Для обнаружения интронов в изучаемых генах клонированный в составе геномной ДНК ген гибридизуют со зрелой РНК, кодируемой этим геном. При наличии в гене интронов их последовательности в ДНК-РНК-гибридах освобождаются в виде одноцепочечных участков и могут быть специфически гидролизованы S1-нуклеазой. При электрофоретическом разделении продуктов гидролиза в денатурирующих условиях при наличии интронов наблюдают появление фрагментов ДНК, причем на основании размеров фрагментов можно локализовать положение интронов в гене. Такой же подход используется и для анализа гомологии между двумя последовательностями нуклеиновых кислот или обнаружения мутаций.
|
|
|
Использование S1-нуклеазы позволяет с точностью до одного–двух нуклеотидов картировать положение точек инициации и терминации транскрипции внутри гена. И в этом случае короткие меченые фрагменты 5’- или 3’-концевых частей гена, заключающие в себе указанные последовательности ДНК, гибридизуют с исследуемыми РНК и образовавшиеся ДНК-РНК-гибриды инкубируют с S1-нуклеазой. Электрофоретическая подвижность полученного фрагмента ДНК, защищенного от действия S1-нуклеазы концевой частью анализируемой РНК, сравнивается с таковой фрагментов того же участка ДНК, образующихся при его секвенировании по методу Максама-Гилберта. В результате однозначно определяют положение нуклеотидов во фрагментах исследуемых ДНК, которые защищены в ДНК-РНК-гибридах от действия S1-нуклеазы.
Футпринтинг
Принцип защиты последовательности нуклеотидов рестрикционных фрагментов ДНК белками от действия агентов, расщепляющих ДНК, лежит в основе футпринтинга – метода, позволяющего определять места специфических контактов белков с ДНК. Этот метод оказался особенно полезным для точной локализации последовательностей нуклеотидов генов, взаимодействующих с регуляторными белками, активаторами и репрессорами, а также с самими РНК-полимеразами, и своему появлению целиком обязан генной инженерии. Рестрикционные фрагменты ДНК, последовательности нуклеотидов которых известны, метят по одному из концов 32P и инкубируют с исследуемыми белками. Образовавшиеся комплексы белок–ДНК подвергают действию агентов, гидролизующих ДНК, например ДНКазы I, в условиях неполного расщепления ДНК, и полученные продукты гидролиза разделяют с помощью электрофореза в полиакриламидном геле с последующей авторадиографией. В отсутствие белка на авторадиограммах наблюдают появление полного набора фрагментов ДНК в том виде, как это имеет место при обычном секвенировании ДНК. Участки ДНК, защищенные белком от действия ДНКазы, идентифицируются по исчезновению полос, соответствующих продуктам статистического расщепления ДНК, концы которых попадают в область связывания с белком. При этом на электрофоретических дорожках появляются характерные пропуски (или "следы" – footprints), что и дало название всему методу.
|
|
|


