 |
Культуральные среды и условия роста
|
|
|
|
Культивирование факультативных анаэробов проводили на 1.5% плотной среде THB (Difco, США) с добавлением 0.5% дрожжевого экстракта (Helicon, Россия) и 5% крови барана при температуре 37°С и 5% СО2 в течение 18 часов.
Выделение чистой культуры
Рассев исходного биологического материала производили методом истощающего штриха (по Дригальски). Метод истощающего штриха предполагает высев культуры на поверхность агаризованной среды в чашку Петри. На первом этапе петлей с культурой наносят ряд параллельных штрихов на агаризованной среде (рис.2.3.1, А). Петлю стерилизуют, остужают о незасеянную часть агаризованной среды и проводят серию штрихов в направлении, перпендикулярном первым (рис.2.3.1, Б). Затем петлю вновь стерилизуют, остужают и штрихи наносят в направлении В (рис.2.3.1), а после очередной стерилизации – в направлении Г (рис.2.3.1). Чашку помещают в термостат и через определенное время учитывают результаты. Обычно на штрихах А и Б вырастает большое число колоний (иногда сплошной рост), тогда как на штрихах В и Г формируются изолированные колонии (http://studopedia.ru/7_23671_metodi-videleniya-chistoy-kulturi.html).
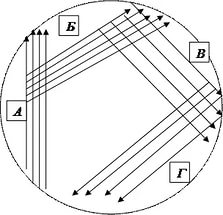
Рис.2.3.1 Метод истощающего штриха
Выделение тотальной ДНК из исходного биологического материала
Тотальную ДНК из исходного биологического материала выделяли с помощью тест-системы для ПЦР «ДНК-экспресс» (Литех, Россия) в соответствии с инструкцией.
Конструирование олигонуклеотидных праймеров
Конструирование, анализ олигонуклеотидных праймеров и определение температуры плавления праймеров осуществляли с помощью компьютерных программ Primer 3 и OLIGO 4.0 (Таблица 2.3.1).
Таблица 2.3.1
Олигонуклеотидные праймеры S.sanguinis и S.gordonii
| Название | 5’→3’ | Тотж. | Размер фрагмента (п.н.) | |
| S.sanguinis | ||||
| Sang1 | GGATAGTGGCTCAGGGCAGCCAGTT | 59,7 | ||
| Sang2 | GAACAGTTGCTGGACTTGCTTGTC | |||
| S.gordonii | ||||
| Gord1 | TGCTTTTCCACTCGACTCTCTC | 55,4 | ||
| Gord2 | TCCTGGAGCAAATTGATCTTGT | |||
| UniBF | AGAGTTTGATCCTGGCTCAG | 55,4 | ||
| UniBR | GGACTACCAGGGTATCTAAT | 51,9 |
|
|
|
Полимеразная цепная реакция (ПЦР)
Полимеразная цепная реакция (ПЦР, PCR) – это метод ферментативного получения амплификаций (большого количества копий) исследуемых фрагментов ДНК путем повторных циклов репликации и денатурации (разделения цепи ДНК на отдельные нити). При этом происходит копирование только исследуемого участка ДНК, поскольку только этот участок соответствует заданным условиям и только в том случае, если он присутствует в исследуемом образце (http://doctorspb.ru/articles.php?article_id=1049).
К 0,25 мкг геномной ДНК добавляли 10 мкмолей каждого из специфических праймеров, фланкирующих исследуемую последовательность, буфер с магнием для полимеразы, по 0,2 мМ каждого из 4 дезоксирибонуклеотидтрифосфатов, объем доводили водой до 25 мкл. Добавляли 0,4 мкл термостабильной ДНК полимеразы. На поверхность жидкости наслаивали 40 мкл минерального масла. Пробирки помещали в амплификатор (Терцик, Россия). Смесь инкубировали при t 94 оС в течение 3 минут. Прибор программировали на цикл денатурации 94 oС на 15 секунд, цикл отжига праймеров на 15 секунд, цикл синтеза ДНК 72 oС на 20 секунд. Последовательность таких циклов повторялась 35 раз. После чего смесь инкубировали при t 72 oС в течение 5 минут. Олигонуклеотидные праймеры, использованные в работе, приведены в таблице 2.3.1.
Электрофорез и выделение ДНК из агарозного геля, определение концентрации ДНК
Электрофорез ДНК проводили в 1,0% агарозном геле в горизонтальном аппарате «Hoefer HE 33» (Pharmacia, Швеция) с использованием ТАЕ буфера. Время электрофореза – 30 мин, напряжение устанавливали 70В. Для визуализации ДНК в ультрафиолетовых лучах в гель добавляли раствор бромистого этидия (0,5 мкг/мл). Визуализацию результатов электрофореза проводили в ультрафиолетовом свете с использованием системы видеозахвата «Versa Doc MP 4000» (Bio Rad) и системы видеозахвата, использующей цифровой фотоаппарат (Olimpus, Япония) и компьютерную программу «Quantity One» (США).
|
|
|
Для расчета молекулярных масс исследуемых фрагментов ДНК использовали ДНК-маркер «100 bp Plus DNA ladder».
Выделение ДНК из агарозного геля осуществляли с использованием коммерческого набора "QIAquick Gel Extraction Kit" (Qiagen, США), согласно прилагаемой инструкции.
Концентрацию ДНК в пробах измеряли с помощью флюориметра «Qubit» с использования набора для измерений «Quant-iTTM dsDNA BR Assay kit, 100 assays *2-1000 ng*».
|
|
|


