 |
R. Muc 13 tac-catgcagtcgacgatgaagcctagcttgctaggtggattagtggcgaacgggtgag 71 1211 71 taatacgtgagtaacctacctttaactctgggataagcctgggaaactgggtctaatacc 130
|
|
|
|
R.muc 72 TAATACGTGAGTAACCTACCTTTAACTCTGGGATAAGCCTGGGAAACTGGGTCTAATACC 131 1211 131 GGATACGACCAATCTCCGCATGGGGTGTTGGTGGAAAGCGTTATGTAGTGGTTATAGATG 190
R.muc 132 GGATACGACCAATCTCCGCATGGGGTGTTGGTGGAAAGCGTTATGTAGTGGTTATAGATG 191 1211 191 GGCTCACGGCCTATCAGCTTGTTGGTGAGGTAACGGCTCACCAAGGCGACGACGGGTAGC 250
R.muc 192 GGCTCACGGCCTATCAGCTTGTTGGTGAGGTAACGGCTCACCAAGGCGACGACGGGTAGC 251 1211 251 CGGCCTGAGAGGGTGACCGGCCACACTGGGACTGAGACACGGCCCAGACTCCTACGGGAG 310
R.muc 252 CGGCCTGAGAGGGTGACCGGCCACACTGGGACTGAGACACGGCCCAGACTCCTACGGGAG 311 1211 311 GCAGCAGTGGGGAATATTGCACAATGGGCGCAAGCCTGATGCAGCGACGCCGCGTGAGGG 370
R.muc 312 GCAGCAGTGGGGAATATTGCACAATGGGCGCAAGCCTGATGCAGCGACGCCGCGTGAGGG 371 1211 371 ATGACGGCCTTCGGGTTGTAAACCTCTGTTAGCAGGGAAGAAGAGAGATTGACGGTACCT 430
R.muc 372 ATGACGGCCTTCGGGTTGTAAACCTCTGTTAGCAGGGAAGAAGAGAGATTGACGGTACCT 431 1211 431 GCAGAGAAAGCGCCGGCTAACTACGTGCCAGCAGCCGCGGTAATACGTAGGGCGCGAGCG 490
R.muc 432 GCAGAGAAAGCGCCGGCTAACTACGTGCCAGCAGCCGCGGTAATACGTAGGGCGCGAGCG 491 1211 491 TTGTCCGGAATTATTGGGCGTAAAGAGCTTGTAGGCGGTTTGTCGCGTCTGCTGTGAAAG 550
R.muc 492 TTGTCCGGAATTATTGGGCGTAAAGAGCTTGTAGGCGGTTTGTCGCGTCTGCTGTGAAAG 551 1211 551 GCCGGGGCTTAACTCCGTGTATTGCAGTGGGTACGGGCAGACTAGAGTGCAGTAGGGGAG 610
|
R.muc 552 GCCGGGGCTTAACCCCGTGTATTGCAGTGGGTACGGGCAGACTAGAGTGCAGTAGGGGAG 611 1211 611 ACTGGAATTCCTGGTGTAGCGGTGGAATGCGCAGATATCAGGAGGAACACCGATGGCGAA 670
R.muc 612 ACTGGAATTCCTGGTGTAGCGGTGGAATGCGCAGATATCAGGAGGAACACCGATGGCGAA 671 1211 671 GGCAGGTCTCTGGGCTGTAACTGACGCTGAGAAGCGAAAAGCATGGGGAGCGAACAGGAT 730
R.muc 672 GGCAGGTCTCTGGGCTGTAACTGACGCTGAGAAGCG-AAAGCATGGGGAGCGAACAGGAT 730 1211 731 TAGATACCCTTGGT 744
|
R.muc 731 TAGATACCCT-GGT 743 Рис. 3.3.9Результат сравнения сиквенсов для культуры 1211 и Rothia mucilaginosa, штамм TeTT Результаты идентификации выделенных пяти культур с использованием секвенирования ДНК-фрагментов, кодирующих 16S рибосомальную РНК, представлены в таблице 3.3.4.
Таблица 3.3.4
Идентификация выделенных культур с помощью секвенирования ДНК-фрагментов, кодирующих 16S рибосомальную РНК
|
|
|
| № | Чистая культура | Название микроорганизма | Степень идентификации |
| Rothia mucilaginosa | 99% | ||
| Rothia mucilaginosa | 99% | ||
| Streptococcus mitis | 99% | ||
| Streptococcus mitis | 99% | ||
| Streptococcus oralis | 99% |
Анитибиотикочувствительность идентифицированных чистых культур
Результаты антибиотикочувствительности идентифицированных чистых культур представлены на рис. 3.4.1 и в таблицах 3.4.1 и 3.4.2

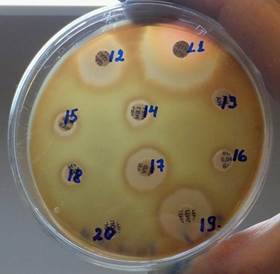
Рис. 3.4.1 Результаты антибиотикочувствительности культуры 531- Streptococcus mitis
На основании результатов антибиотикочувствительности в приведенных ниже таблицах 3.4.1 и 3.4.2 можно сделать выводы, что все идентифицированные нами микроорганизмы чувствительны к Азитромицину (относительно новый препарат из группы макролидов на Российском рынке) и практически все микроорганизмы чувствительны к антибиотикам из группы бета-лактамов, кроме Streptococcus sanguinis, Neisseria perflava и некоторых штаммов Rothia mucilaginosa. Также эти микроорганизмы устойчивы к препарату из группы фторхинолонов Ципрофлоксацину, к которому в свою очередь чувствительны остальные микроорганизмы. Наибольшую устойчивость микроорганизмы проявили к полимиксинам, аминогликозидам и полипептидам.
Таблица 3.4.1
Антибиотикочувствительность идентифицированных чистых культур
| М/О | АНТИБИОТИКИ | ||||||||||
| Бензил-Пенициллин | Оксациллин | Ампициллин | Карбенициллин | Пиперациллин | Цефазолин | Цефамандол | Цефоперазон | Цефотаксим | Цефтазидим | Цефепим | |
| R.mucilaginosa | у | у | у | ч | ч | п | ч | ч | ч | п | ч |
| R.mucilaginosa | у | ч | у | ч | - | ч | ч | п | п | п | п |
| S.gordonii | ч | ч | ч | ч | - | ч | ч | ч | ч | ч | ч |
| S.anginosus | ч | ч | ч | ч | - | ч | ч | ч | ч | п | ч |
| S.mitis | ч | ч | ч | ч | - | ч | ч | ч | ч | ч | ч |
| S.sanguinis | у | у | у | п | ч | у | п | у | п | п | п |
| S.oralis | ч | ч | ч | ч | - | ч | ч | ч | ч | ч | ч |
| S.sanguinis | у | ч | у | ч | - | п | п | у | п | ч | п |
| S.mitis | ч | ч | ч | ч | - | ч | ч | ч | ч | ч | ч |
| N.perflava | у | у | у | ч | ч | ч | ч | ч | п | ч | ч |
| S.orlis | ч | ч | ч | ч | ч | ч | ч | ч | ч | ч | ч |
| R.mucilagenosa | ч | ч | ч | ч | ч | ч | ч | ч | ч | ч | ч |
| R.mucilagenosa | ч | ч | ч | ч | ч | ч | ч | ч | ч | ч | ч |
|
|
|
· Ч – чувствительны
· П – промежуточно чувствительны
· У - устойчивы
Таблица 3.4.1
Антибиотикочувствительность идентифицированных чистых культур
| М/О | АНТИБИОТИКИ | |||||||||
| Имипенем | Меропенем | Ванкомицин | Азитромицин | Ципрофлоксацин | Полимиксин | Канамицин | Гентамицин | Амикацин | Бацитрацин | |
| R.mucilaginosa | ч | ч | ч | - | у | - | у | у | у | у |
| R.mucilaginosa | п | п | ч | п | у | ч | у | у | у | у |
| S.gordonii | ч | ч | ч | - | ч | у | у | п | у | у |
| S.anginosus | ч | ч | ч | п | п | ч | у | у | у | у |
| S.mitis | ч | ч | ч | ч | ч | ч | у | ч | у | у |
| S.sanguinis | ч | ч | ч | - | у | - | у | у | у | у |
| S.oralis | ч | ч | ч | ч | ч | ч | у | п | у | у |
| S.sanguinis | п | ч | ч | - | п | у | у | п | у | у |
| S.mitis | ч | ч | ч | - | п | у | у | у | у | у |
| N.perflava | ч | ч | у | - | у | - | п | ч | п | у |
| S.orlis | ч | ч | ч | - | п | - | у | у | у | у |
| R.mucilagenosa | ч | ч | ч | ч | ч | у | п | - | ч | - |
| R.mucilagenosa | ч | ч | ч | ч | ч | ч | у | - | у | - |
· Ч – чувствительны
· П – промежуточно чувствительны
· У – устойчивы
|
|
|


